Dados LiDAR do GeoSampa (do .LAZ ao .TIF)
Trabalhando com dados LiDAR no R
 MDS com sobreposição de pontos das arvores mais altas por janelas de 5 x 5 m
MDS com sobreposição de pontos das arvores mais altas por janelas de 5 x 5 m
No primeiro post sobre como baixar os dados LiDAR da plataforma GeoSampa em lote, acabei não entrando em detalhes sobre o que é o LiDAR, assumindo que quem está baixando já sabe o que é. Mas neste, antes de ir para o principal que é: processamento e transformação de nuvens de pontos LiDAR para o formato raster (.tif), só para dar uma relembrada, vou iniciar a conversa abordando o que é o LiDAR.
LiDAR, o que é?!
Penso que eu não chego no LiDAR sem falar um pouco de Sensoriamento Remoto, mesmo que seja em um parágrafo. Sensoriamento Remoto, numa definição simples, é a aquisição de dados sobre objetos sem ter contato físico com eles. A aquisição deste dado é feita através de sensores instalados a bordo de alguma plataforma que pode ser de nível orbital, aéreo ou terrestre. Geralmente, escutamos falar sobre imagens de satélites, na verdade, os satélites carregam sensores que realizam esta aquisição de dados remotamente que após processados se tornem essas imagens de observação da Terra ou outros objetos que costumamos ver. Até aqui, já vimos dois elementos importantes, o sensor e o alvo (objeto a ser detectado), mas tem mais dois para fechar com os quatro elementos principais deste sistema. Os outros dois são a Radiação Eletro Magnética(REM) e a Fonte. Para fazer simples, o Sensor capta/coleta a REM refletida pelo Alvo vindo de alguma Fonte. Podemos ver que a REM é o elemento de ligação entre os três outros. No ponto de vista da fonte da REM que incide o objeto/alvo, que depois, a sua reflexão será captada pelo sensor, os sensores podem ser classificados em sensores ativos (quando o sensor também é a fonte da REM) e passivos (quando a fonte da REM incidente não é o sensor, ex: o Sol; pode ser também a Terra para os sensores passivos de micro-ondas e termais).
Agora vamos para o LiDAR que deve ficar mais natural. O LiDAR (Light Detection and Ranging) - em tradução literal para o português: detecção e estimativa da distância pela luz - é um sistema de sensoriamento ativo. Como visto anteriormente, o sistema ativo gera sua própria energia - neste caso, a luz - para medição de objetos. Em um sistema LiDAR, a luz é emitida por um laser de disparo rápido, operado no infravermelho próximo em um comprimento de onda em torno de 900 nm. Essa luz viaja para o chão e reflete em coisas como árvores, água, prédios e outras estruturas construídas. A energia da luz refletida retorna ao sensor LiDAR onde é registrada. Com base no tempo de viagem bidirecional da energia do laser e na velocidade da luz, pode-se calcular um alcance até o ponto de reflexão. Com o conhecimento da posição e orientação precisas da aeronave, a direção do feixe de laser e o alcance, o objeto que produziu o reflexo pode ser geolocalizado com precisão em três dimensões.
A cobertura do solo, como a vegetação, normalmente reflete uma parte da energia do laser do topo do dossel, enquanto a energia residual pode refletir ainda mais do dossel inferior, do sub-bosque e do solo sob a vegetação, i.e. várias reflexões podem ser registradas a partir de um pulso de luz. Portanto, o LiDAR tem uma capacidade única de caracterizar a estrutura tridimensional das florestas, bem como informações sobre a superfície do solo sob a vegetação.
Curiosidade: LiDAR vs RADAR vs SONAR
LiDAR, RADAR e SONAR são Light Detection And Ranging, Radio Detection And Ranging e Sound Navigation and Ranging, respetivamente. É muito comum a gente se perguntar qual a diferença entre essas três tecnologias. Sob certos ângulos, elas compartilham a mesma ideia, que é a de detecção de objetos e estimativa de distância. Embora tenham objetivos sobrepostos, a simples mudança de tipo de onda usada torna suas capacidades muito diferentes umas das outras. Por um lado, o LiDAR e o RADAR fazem essa detecção e medição utilizando REM de comprimento de ondas diferentes, no caso luz/laser e ondas de rádio respectivamente. O SONAR, utiliza ondas sonoras (existem duas tecnologias SONAR, ativo e passivo. O ativo cria um pulso de som, e depois escuta as reflexões/ecos do pulso. O SONAR passivo escuta sem transmitir). Pode ser tentador querer saber qual é a melhor das três tecnologias, mas depende muito da aplicação. Podem até ter sobreposição de uso, mas uma não chega a substituir a outra em utilidades e aplicações. O SONAR é usado principalmente para detectar objetos subaquáticos porque as ondas sonoras podem penetrar nas profundezas da água até o fundo do mar, neste campo os sistemas que usam ondas eletromagnéticas ficam para trás, pois o som se propaga muito melhor na água comparando com as REM. Os sistemas LiDAR devido ao comprimento de ondas eletromagnéticas que utilizam conseguem medir um ambiente com alta precisão e produzir uma imagem 3D com base nos resultados, e por este mesmo fato — o comprimento de onda pequena (entre 750 nm e 1.5µm) — os pulsos LiDAR são afetados negativamente pelas condições climáticas e atmosféricas, como neblina densa, fumaça e até chuva. A tecnologia RADAR ao contrário do LiDAR, não é afetada por condições climáticas adversas, como nuvens, chuva ou neblina (assim na navegação aérea mesmo com muitas nuvens um avião vindo será detectado), mas pelo comprimento de ondas que utiliza — maior do que o do LiDAR (entre 3 mm e 30 cm) - não consegue detectar objetos muito pequenos ou fazer uma reprodução 3D do ambiente em torno. É quase uma brincadeira “pedra, papel e tesoura”. Em vez de qual é melhor, os sistemas LiDAR, RADAR e SONAR têm seu próprio lugar com suas vantagens e desvantagens.
Mas vamos voltar ao LiDAR, o assunto deste post…bora!
Usos do LiDAR nas geotecnologias
A técnica LiDAR é utilizada principalmente para levantamentos topográficos de alta resolução, para caracterizar a estrutura da vegetação (altura e cobertura do dossel), bem como a volumetria de construções e ambientes urbanos de forma mais rápida e confiável.
O LiDAR permite gerar produtos como o Modelo Digital de Terreno (MDT) representando o terreno sem nenhuma cobertura, e o Modelo Digital de Superfície (MDS) representando os edifícios, árvores, etc. São estes dois produtos que se encontram disponíveis na plataforma do GeoSampa.
Formato dos arquivos
Na maioria das vezes os dados LiDAR estão disponíveis como pontos discretos (existe a opção de onda completa também). Uma coleção de pontos LiDAR de retorno discreto é conhecida como nuvem de pontos LiDAR. O formato de arquivo comumente usado para armazenar nuvem de pontos LiDAR é o .las. O formato .laz é uma versão altamente compactada do .las e está se tornando mais amplamente utilizado.
Atributos de dados LiDAR
Os atributos de dados LiDAR podem variar, dependendo de como os dados foram coletados e processados. Você pode descobrir quais atributos estão disponíveis para cada ponto LiDAR observando os metadados. Todos os pontos de dados LiDAR terão uma localização X, Y associada e valor Z (elevação). A maioria dos pontos de dados LiDAR terá um valor de intensidade, representando a quantidade de energia luminosa registrada pelo sensor. Alguns dados LiDAR também serão “classificados” com especificações sobre o que são os dados. A classificação de nuvens de pontos LiDAR é uma etapa de processamento adicional. A classificação simplesmente representa o tipo de objeto do qual o retorno do laser é refletido. Portanto, se a energia da luz refletida é de uma árvore, pode ser classificada como “vegetação”. E se refletido no chão, pode ser classificado como “solo”. Alguns produtos LiDAR são classificados de forma binária como “solo ou não-solo”. Alguns outros podem ser mais detalhados e classificados conforme o tipo de vegetação. As classes são definidas pela ASPRS(the American Society for Photogrammetry and Remote Sensing). Os valores das classes são numéricos, e estão geralmente entre 0 e 20 mas podem assumir valor até superior. Algumas classes mais comuns incluem:
- 0 Nunca classificado
- 1 Não atribuído
- 2 Chão
- 3 Vegetação (baixa)
- 4 Vegetação (média)
- 5 Vegetação (alta)
- 6 Construções
- 9 Água
Para mais detalhes a respeito das classes, podem dar uma olhada nessas duas páginas: página 1, página 2.
Área de interesse
Na plataforma GeoSampa são disponibilizados dois produtos LiDAR, nuvem de pontos MDT e MDS obtido por aerolevantamento em 2017, com precisão da ordem de 10 cm. Aqui estarei utilizando o MDS com o objetivo de explorar melhor as opções de processamento, visto que podemos chegar ao MDT pela nuvem do MDS.
Assumo que você já tem as quadrículas relacionadas à sua área de trabalho, no seu disco local. Para esta demostração, eu baixei 15 quadriculas que correspondem a parte da cidade universitária da Universidade de São Paulo (Campus Capital). Utilizei uma seleção por atributo espacial conforme mostrado no final do post anterior utilizando gdal e sqlite diretamente no bash.
O bash script direto, para quem não quer ir lá rs
|
|
Ferramentas de processamento
Para trabalhar com a nuvem de pontos LiDAR usarei a linguagem R, com ajuda do pacote lidR. Este pacote permite ler e escrever arquivos .las e .laz, plotar nuvens de pontos, conversão de formatos e muitos outros processamentos relacionados a nuvem de pontos LiDAR em geral. Se você trabalha diretamente com isso é importante dar uma olhada na documentação deste pacote que a galera disponibiliza gratuitamente, inclusive um livro meio que em formato de tutorial The lidR package, confesso que aprendi muito com este material.
Uma coisa que eu gostei demais deste pacote, é que ele já permite o processamento de arquivos grandes demais para a nossa RAM por meio de processamento por chunks (pedaços) e em paralelo via o LAScatalog processing engine. Ao trabalhar com dados LiDAR, vai se deparar com nuvens de pontos muito densas, numa brincadeira simples a gente pode ter que processar bilhões de pontos, aí o processador engasga e o sistema simplesmente trava e/ou desliga. Costumo brincar que o R é “single core”, mas a gente não é obrigado a usar só um “core” se investimos em uma máquina com processador “multi-cores” (multi-núcleos), mas isso vem a um custo, você precisa transformar a sua rotina para rodar em paralelo e usar quantas cores que tem direito…voltarei a explorar este ponto em específico em outro post. Aqui, o lidR já faz tudo para nós, facilitando a computação em paralela, e isso é ótimo!
Explorando os dados
Vamos ver os arquivos que eu baixei para a brincadeira aqui:
|
|
>>> [1] "3313-151.laz" "3313-152.laz" "3313-153.laz" "3313-154.laz" "3313-161.laz"
>>> [6] "3313-162.laz" "3313-163.laz" "3313-164.laz" "3313-241.laz" "3313-243.laz"
>>> [11] "3313-321.laz" "3313-322.laz" "3313-331.laz" "3313-332.laz" "3313-411.laz"
Como se pode ver, estou com 15 quadrículas/arquivos .laz.
Antes de tudo, vamos carregar o pacote lidR
|
|
Processamento de um arquivo
|
|
>>> class : LAS (v1.0 format 1)
>>> memory : 386.6 Mb
>>> extent : 323507.8, 324050.5, 7393143, 7393730 (xmin, xmax, ymin, ymax)
>>> coord. ref. : NA
>>> area : 324416 units²
>>> points : 6.33 million points
>>> density : 19.52 points/units²
>>> density : 15.61 pulses/units²
Sim, você viu bem, são mais de 6 milhões de pontos, com uma densidade de 19 pontos por unidade de área, e isso vem de um arquivinho de 26 MB. Podemos ver que a descrição não fala qual é a unidade de área, isso porque o pacote não sabe qual é o sistema de coordenada dos dados carregados. Ainda bem que isso é informado lá na plataforma. Eles estão no datum SIRGAS 2000, sistema UTM na Zona 23 S, vamos fazer a associação.
|
|
>>> class : LAS (v1.0 format 1)
>>> memory : 386.6 Mb
>>> extent : 323507.8, 324050.5, 7393143, 7393730 (xmin, xmax, ymin, ymax)
>>> coord. ref. : SIRGAS 2000 / UTM zone 23S
>>> area : 321048 m²
>>> points : 6.33 million points
>>> density : 19.73 points/m²
>>> density : 15.78 pulses/m²
Agora sim, temos informações sobre as unidades! A área total é em torno de 32 ha ou 0.32 km²…
Quais classes estão presentes nesta nuvem de pontos?
|
|
>>> [1] 19 2 20 5 6
Nesta quadrícula, estão presentes as classes 19, 2, 20, 5 e 6. Podem dar uma verificada nas duas páginas que eu passei aqui em cima, mas vamos filtrar e dar uma olhada em algumas classes e ver o que representam.
select e filter, para escolher somente os atributos necessários e assim poupar memoria (veja a documentação da função rodando ?readLAS no console para saber as opções disponíveis).
Visualização de nuvens de pontos
O pacote lidR permite plotar nuvens de pontos, faremos isso utilizando um filtro para as classes que queremos.
Chão
|
|

Construção
|
|

Vegetação
|
|

Transformando para raster
As nuvens de pontos fornecem muitas informações, no entanto, trabalhar com elas pode ser difícil devido ao tamanho dos arquivos, a memória por elas ocupada ao carregá-las numa seção de trabalho e as ferramentas disponíveis para LiDAR com grandes volumes de pontos. Por isso, os produtos/dados LiDAR são geralmente transformados em formato de grid ou raster. Visto que o formato raster é suportado por muitos programas comumente usados assim pode ser mais fácil para mais pessoas trabalharem.
Um arquivo raster é composto de uma grade regular de células que são renderizados em um mapa como pixels, todos do mesmo tamanho. Cada valor de pixel representa uma área na superfície da Terra (se tratando de dados de observação da terra). Essa área é definida pela resolução espacial do raster.
O pacote lidR oferece três funções para rasterizar, rasterize_terrain(), rasterize_canopy() e rasterize_density(). A primeira interpola os pontos classificados como chão e água e produz um raster de MDT, a segunda função cria um MDS usando diversos algoritmos enquanto a terceira cria um raster de densidade dos pontos. Diversos algoritmos que essas funções utilizam já estão disponibilizados pelo pacote mesmo, como: TIN e IDW para MDT, p2r e pitfree para MDS. No âmbito de uma pesquisa, será necessário justificar a escolha de um interpolador/algoritmo ou outro. Na documentação do pacote, aqui, apresenta algumas vantagens e desvantagens relacionas a cada escolha e algumas boas práticas.
Apresentarei três testes de rasterização, um criando um raster de MDT, e dois criando raster de MDS representando o dossel (vegetação) e as construções.
MDT (raster)
|
|
>>> class : SpatRaster
>>> dimensions : 588, 544, 1 (nrow, ncol, nlyr)
>>> resolution : 1, 1 (x, y)
>>> extent : 323507, 324051, 7393142, 7393730 (xmin, xmax, ymin, ymax)
>>> coord. ref. : SIRGAS 2000 / UTM zone 23S (EPSG:31983)
>>> source : memory
>>> name : Z
>>> min value : 720.38
>>> max value : 745.27
Já estamos com um raster de MDT! A partir daqui, pode exportá-lo como arquivo .tif e utilizar no seu fluxo de processamento. Mas aqui vamos plotar e ver como se parece.
|
|
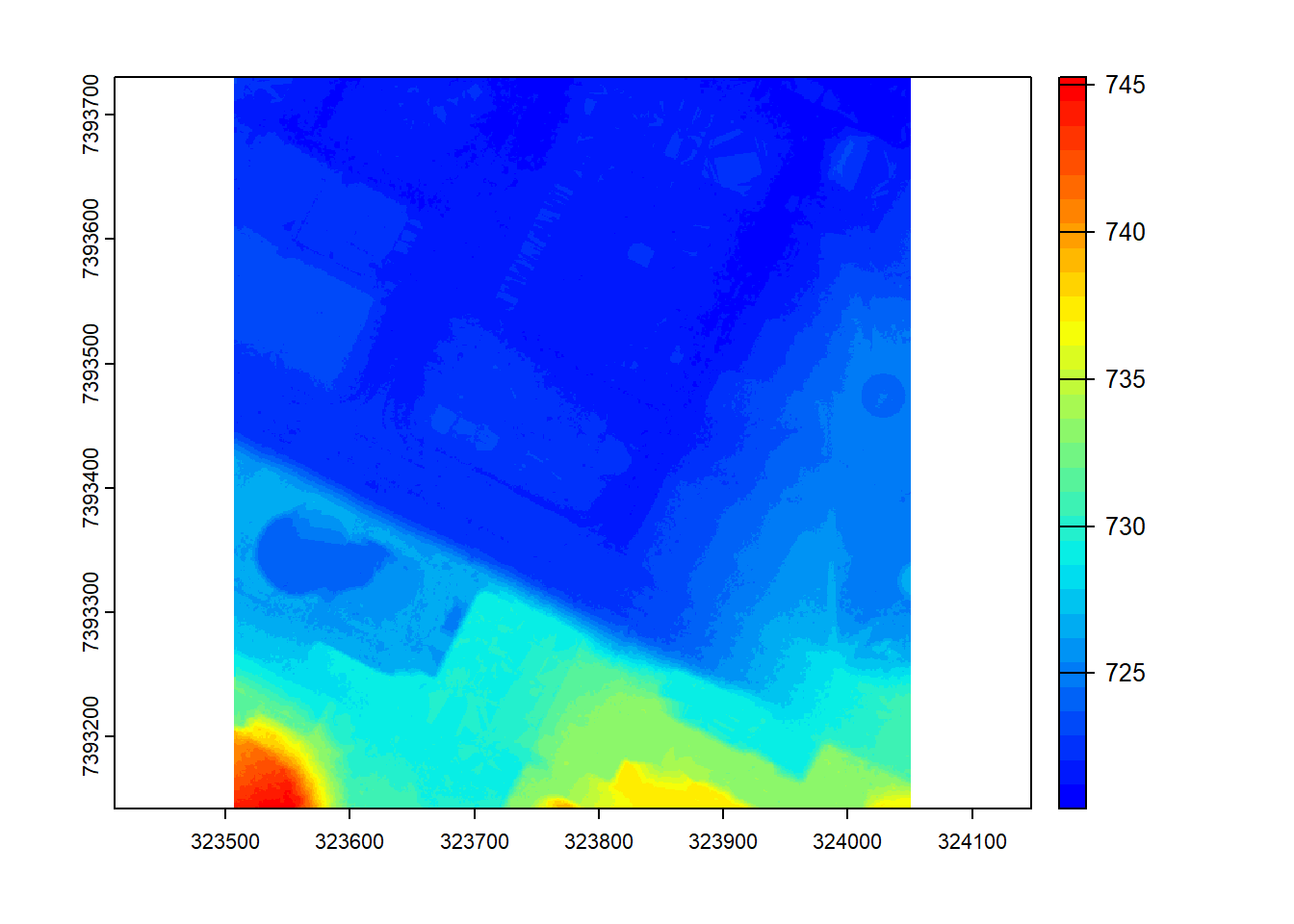
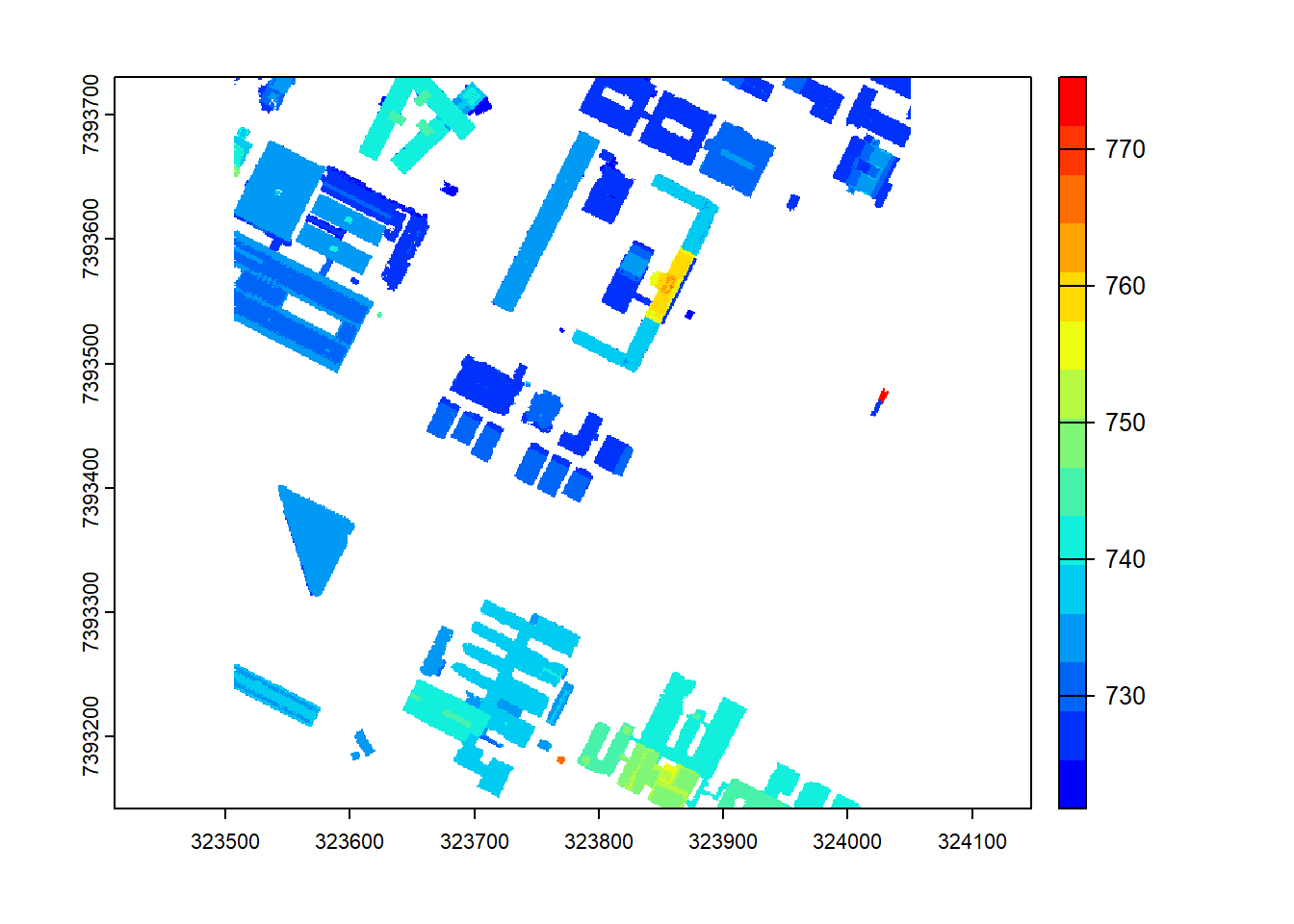
MDS construções (raster)
|
|
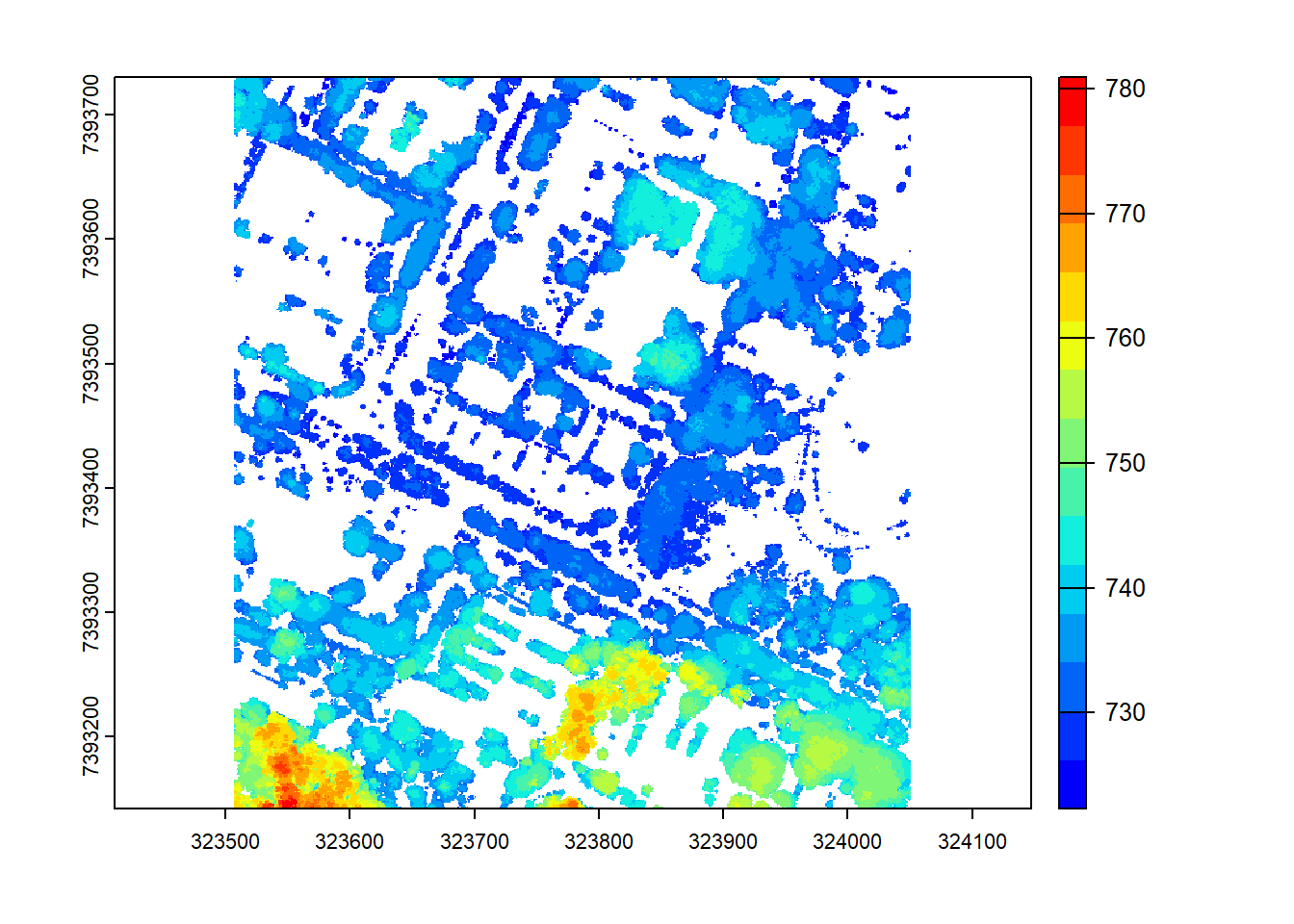
MDS vegetação (raster)
|
|
Outra função bem interessante, é a normalização das alturas com normalize_height(), aplicável para MDS como as vegetações e construções. Este procedimento “elimina” o efeito da variação topográfica do terremo, fazendo com que as alturas começam na mesma referência (mesmo ponto zero), e assim tornar possível comparar a altura de uma árvore com a outra ou de um prédio com outro mesmo numa distância considerável. O pacote tem muuuita coisa, que não consigo tocar cada um neste post (senão acabarei escrevendo a versão portuguesa do livro rsrs), então vale muito a pena dar uma olhadinha por lá.
Processamento de coleção de arquivos
Na primeira parte de processamento, trabalhamos com só um arquivo, mas lembra que eu tenho 15 desses? Então, aqui mostrarei como trabalhar com mais de um arquivo, da melhor forma.
Podemos pensar que é só fazer uma iteração sobre todos os arquivos e criar a saída necessária para depois concatenar tudo criando um mosaico de sua área. O problema disso, é que os efeitos de borda vão ficar muito evidente em cada junção, assim criando um monte de traços indesejados no meio, pois cada quadricula não será processada com um buffer que poderia minimizar, ver eliminar, este efeito. Aí, entra o modulo LAScatalog processing engine para trabalhar com coleção de arquivos permitindo gerenciamento automático de buffer, armazenamento em disco, paralelização dos processos, etc.
|
|
>>> class : LAScatalog (v1.0 format 1)
>>> extent : 322437.4, 325120.7, 7392553, 7394319 (xmin, xmax, ymin, ymax)
>>> coord. ref. : SIRGAS 2000 / UTM zone 23S
>>> area : 4.78 km²
>>> points : 109.51 million points
>>> density : 22.9 points/m²
>>> density : 18.4 pulses/m²
>>> num. files : 15
Se para uma quadrícula, estávamos com uma nuvem de 6 milhões de pontos, agora estamos com uma nuvem de pontos com 109 milhões de pontos, cobrindo uma área de quase 5 km². Imagina carregar as cinco mil quadriculas (5362, para ser exato) do município de São Paulo aí…eu sei do MDT, pois já precisei carregar isso, deu 1.13 bilhão de pontos (para MDS deve ser muito mais de que isso ainda), sem o LAScatalog processing engine não conseguiria processar tudo isso. Mas aqui, vamos com os 15 arquivos mesmo.
Podemos plotar esta coleção (catalog), não a nuvem de ponto, mas a estrutura desta coleção de arquivos. A estrutura é subdividida em chunks (pedaços) e a cada chunk é associada um valor de sobreposição de borda (buffer). O valor padrão do chunk é equivalente a uma quadrícula/arquivo, e o valor padrão do buffer é 30 metros. Na hora de plotar podemos incluir opção de chunk_pattern assim mostrando toda essa estrutura.
|
|
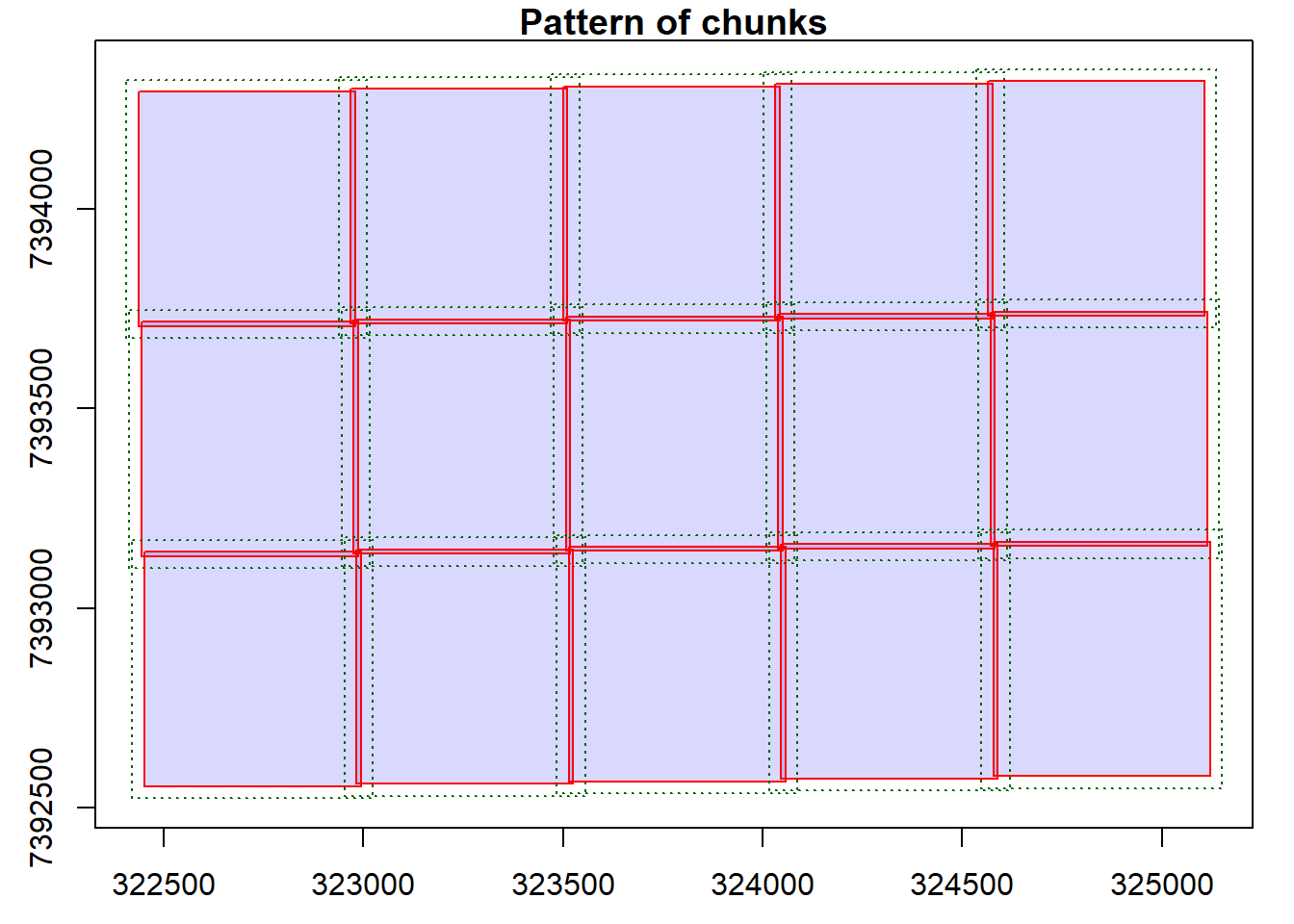
Como isso me ajuda?? Pois bem! com esta estrutura, estamos livres de definir os nossos valores para chunk e/ou buffer. O processamento é feito de um chunk por vez, assim por uma computação paralelizada, podemos processar vários chunks em simultâneo, distribuindo eles nos núcleos que a nossa máquina tem disponíveis. Cada chunk processado é salvo em endereço temporário que pode ser definido pelo usuário, para depois serem concatenados como um arquivo só. E já que este processo foi feito utilizando buffer em todas as bordas, aquele efeito indesejado de borda é eliminado. Uma dica, tenta encontrar o equilíbrio entre a largura dos chunk e a distribuição dos mesmo pelos núcleos que se pode usar, pois, os pontos que devem ser processadas paralelamente devem caber na sua memória RAM. Às vezes é melhor diminuir o tamanho do chunk e distribuir para mais núcleos, pois quanto maior forem os chunks, mais memoria RAM será utilizada, se essa memória não estiver disponível a maquina para de responder e pode até desligar.
Vamos fazer uma configuração para um processamento paralelo aqui.
|
|
Plotando de novo, para ver se algo mudou
|
|
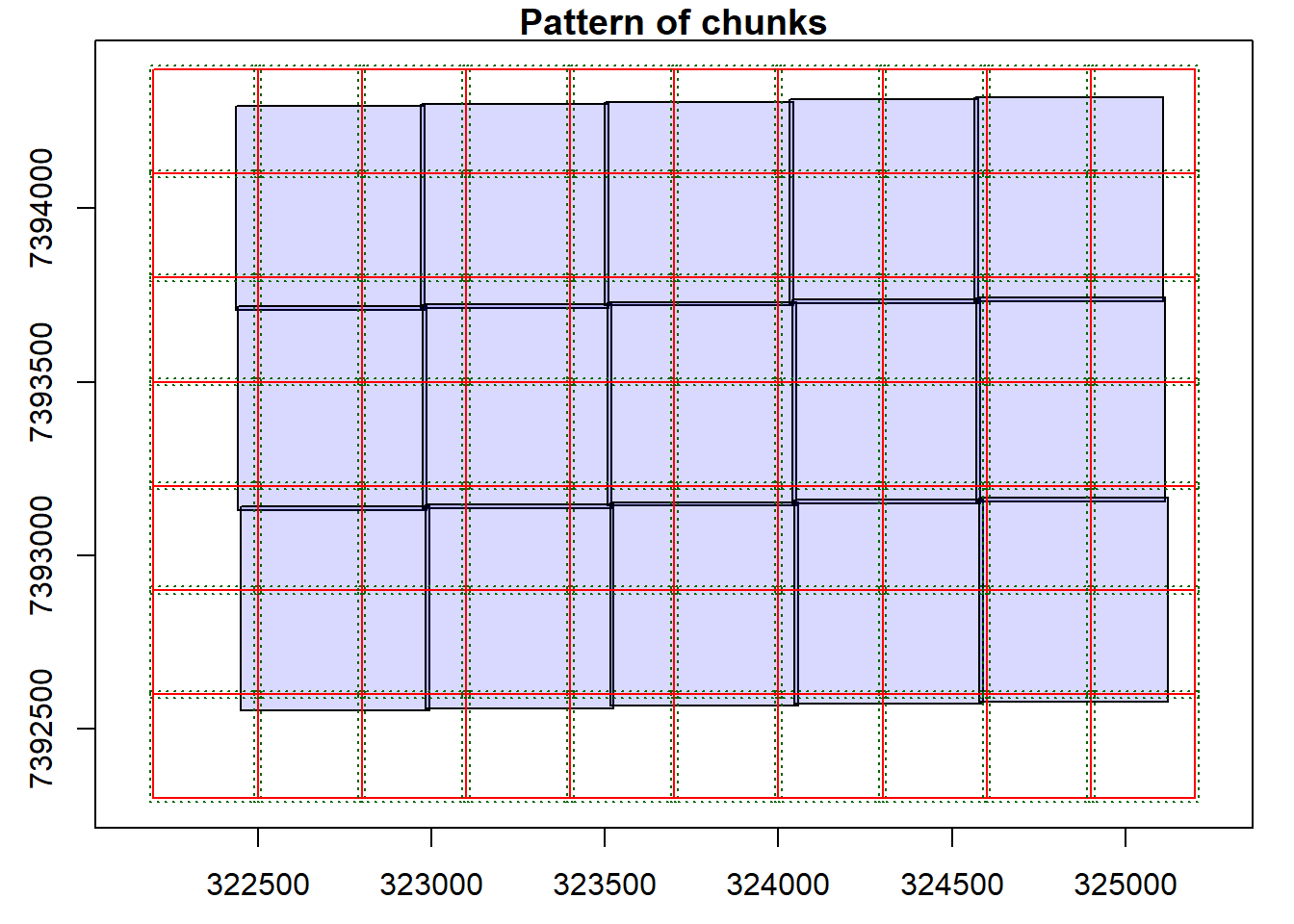
Saímos da situação onde a quantidade de chunk era igual à quantidade de quadrículas/arquivos, agora cara arquivo é dividido em 3 pedaços, i.e., eu consigo processar ~ 6 chunks equivalente a duas quadriculas em 6 núcleos de uma só vez (de novo, se os chunks ficarem muito grandes, a sua RAM acaba antes que os núcleos possam fazer processar aquilo).
Configurando o processamento em paralelo
O pacote lidR, ao ser carregado já está configurado para usar metade dos núcleos disponíveis na máquina para processamento em paralelo (get_lidr_threads() para verificar e set_lidr_threads() para especificar), pois alguns algoritmos já são nativamente paralelizados, são baseados em C++ com OpenMP (Open Multi-Processing). Outra forma de paralelização que o lidR utiliza é por meio do pacote chamado future, permitindo formas do R avaliar expressões de forma assíncrona, na página do pacote explica muito bem os conceitos e como molhar os pezinhos ali.
Com o lidR + future, podemos não somente processar as coisas em paralelo em uma única máquina, mas também colocando vários computadores em rede formando um cluster, aí já entra em computação paralela de forma remota (não cabe aprofundar isso aqui, só para saber que existe).
|
|
Quase pronto, precisamos dizer aonde que queremos salvar os arquivos temporários, resultado de cada chunk processado, senão eles vão sobre carregar a RAM e queremos a RAM livre!
|
|
Rasterizando a coleção
Agora de fato, podemos processar, e ver a mágica acontecer rs…vamos rasterizar um MDT com a função rasterize_terrain(). Mas antes disso, como eu sei que para isso precisarei somente dos pontos de chão e também dos atributos xyz, poderíamos ter carregado somente isso, faz grande diferença para coleções grandes.
|
|

Plotando o arquivo raster final!
|
|

Salvando o raster em disco
Não vamos querer fazer isso toda vez neh, vamos salvar o raster final em disco como um tif, usando o pacote terra.
|
|
Não esperava que o post ficaria tão grande, mas espero que tenha valido a pena ter explorado um pouquinho mais.
Ah, como bonus, aqui abaixo tem o código que gerou a representação 3D no início do post. Nela, além da representação 3D do MDS, é acrescentado pontos das arvores mais altas por janela de 5 m.
Siim, quero ver!
|
|
