Simulando dados de coleta com R
 Based on photo by Geran de Klerk on Unsplash
Based on photo by Geran de Klerk on Unsplash
A ideia principal
Na postagem sobre o Sell Bash apresentei uma situação onde dados sobre algumas espécies de árvores foram coletados e você precisava tratar esses dados e deixá-los de uma forma específica. Era uma pasta com arquivos de várias extensões e as espécies todas misturadas em tabelas e pastas, em fim, bem perto da realidade. Para tratar esses dados, eu precisei primeiro criá-los. No processo de criação desses dados fui tentando deixá-los com uma certa aleatoriedade.
O que precisamos fazer é criar tabelas contendo 5 colunas: espécie, altura, x e y com quantidade de linhas e extensão de arquivos variados, isso distribuído em várias pastas. Existem vários caminhos para realizar esta tarefa, mas aqui estarei mostrando a forma que segui, utilizando a linguagem R.
Os passos podem ser resumidos assim:
- Escolher nomes para cada uma das 5 espécies;
- Criar uma tabela com valor de altura de cada espécie;
- Juntar elas em uma única tabela;
- Bagunçar a ordem das observações;
- Criar um grid de localização;
- Associar cada observação com um ponto no grid, de forma aleatória e sem duplicação;
- Dividir a tabela única em sub-tabelas que podem ser salvas separadamente em pastas diferentes.
Criação de tabelas customizadas
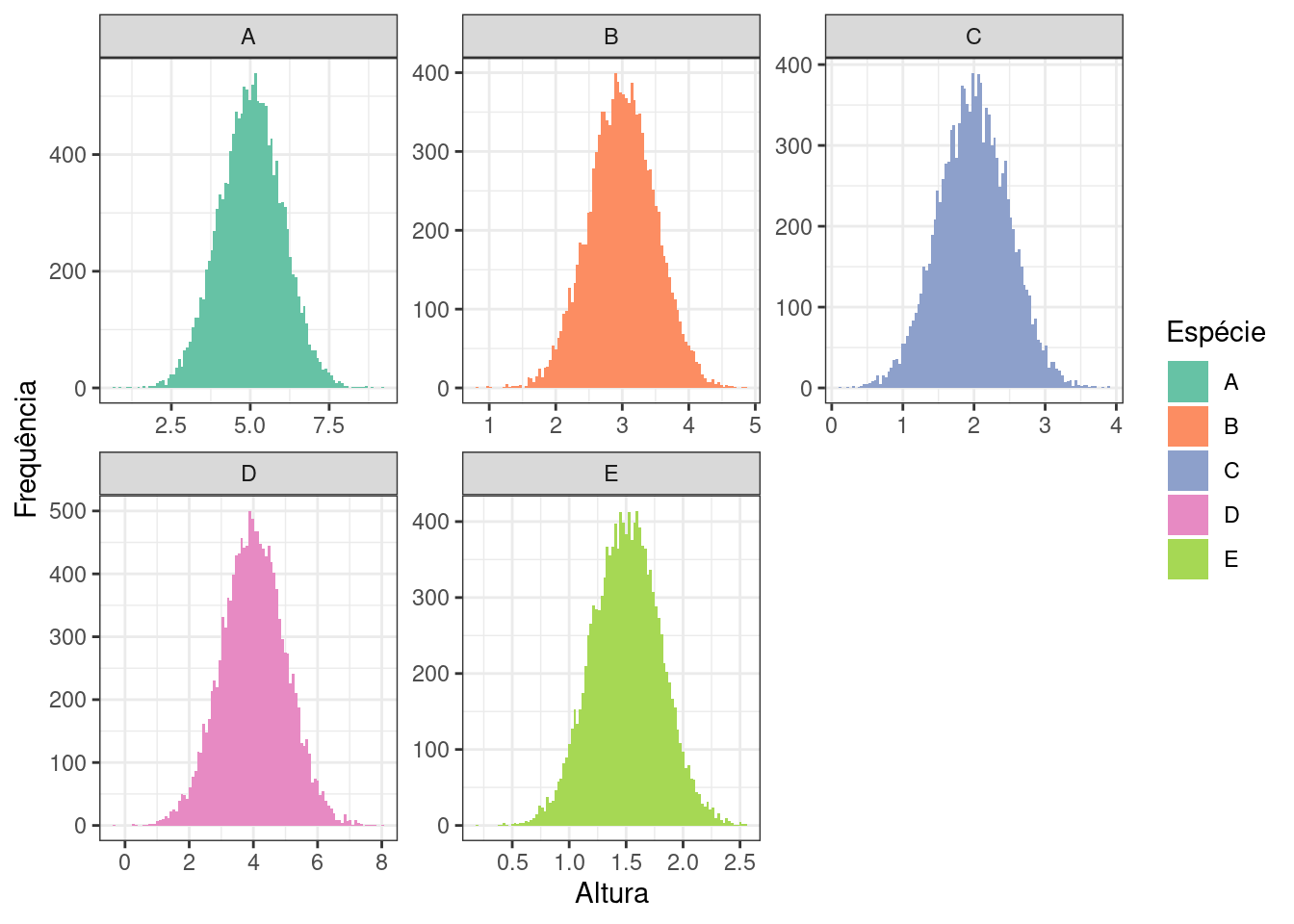
No R temos uma estrutura de dados chamada data frame que é uma estrutura equivalente à tabela. Usarei esta estrutura para criar e salvar as nossas tabelas. Já que eu vou precisar criar uma tabela para cada espécie, vou criar uma função chamada df_maker uma criadora de data frames com alguns parâmetros configuráveis. Também eu gostaria que a variação de altura das observações coletadas seguissem uma distribuição normal, aquela cujo histograma dos dados apresenta um formato de sino (curva Gaussiana), com valor de média e mediana caindo no mesmo lugar e dividindo o sino em dois lados simétricos; e 1, 2 e 3 desvios padrão em volta da média contemplam 68.27%, 95.45% e 99.73% dos dados respetivamente. Escolhi seguir por esta abordagem, pois muita coisa na natureza tendem a apresentar este padrão de comportamento quando observadas o bastante e com uma amostragem feita de forma aleatória.
|
|
A função df_maker() recebe como argumento a média e o desvio padrão, esses dados serão responsáveis para criar uma lista de valores de forma aleatória que seguem uma distribuição normal cuja media e desvio padrão são os valores desses dois parâmetros. Note que, eu escolhi não usar todos os valores, por isso eu sempre crio 10 vezes do que eu preciso e usar 1/10 de forma aleatória. Já que estamos trabalhando com altura escolhi trabalhar somente com 4 dígitos significativos (isso altera o dado um pouco, mas não atrapalha o que estamos fazendo aqui). Agora posso criar a quantidade de tabelas que eu quiser, vamos fazer isso para as 4 espécies A, B, C, D, E.
|
|
Neste bloco, usei a minha função df_maker() já criada, onde eu variei os nomes das espécies e coloquei valor de média e desvio padrão diferentes para cada um. Por estarmos falando de altura de árvores, seria estranho ter alturas negativas, então procurei valores onde a média subtraída por 3 vezes o desvio padrão não desse valor negativo (lembra que 3 desvios padrões +/- a média cobre 99.73% das observações? você saca a ideia rs). Eu abri mão de ter controle sobre quantidade de linhas que cada tabela deveria ter, então essa função set_nrow() resolve este problema para nós, a cada chamada ela entrega um valor entre 10 mil e 15 mil.
Vamos ver como se parece a nossa tabela
|
|
>>> especie altura
>>> 1 A 5.048
>>> 2 A 4.788
>>> 3 A 5.707
>>> 4 A 4.916
>>> 5 A 4.929
>>> 6 A 6.060
Se você reparou bem, como o nome indica, as espécies estão em ordem, podemos bagunçá-los um pouco.
|
|
Agora a tabela se parece assim…
|
|
>>> especie altura
>>> 20828 B 3.708
>>> 23769 B 2.771
>>> 47851 D 4.852
>>> 29663 C 2.733
>>> 16833 B 2.838
>>> 4779 A 5.549
Está faltando algo, não? Sim, precisamos colocar coordenadas x e y para cada observação. Para isso, vou criar um grid seguindo alguns padrões e depois juntar esses dados à nossa primeira tabela. Tem uma função bem interessante para a gente criar combinações deste tipo que é expand.grid() ela recebe dois vetores ou lista de dados e nos retorna todas as combinações entre os seus valores. Para criar esses dados de entrada, estarei supondo que estou em uma zona UTM qualquer onde o ponto mais ao norte é 10 mil quilômetros (o valor da linha do equador quando estamos no hemisfério sul, estando no hemisfério norte ela vale zero) e sabendo que o meridiano central de cada zona dessas vale 500 quilômetros usaremos esses dados como ponto de partida. Então temos:
|
|
Eu quero 10 km ao sul deste zero norte e 10 km ao oeste deste zero este, agora é só subtrair, assim teremos um quadrado de 100 km2. Para criar um grid que cobre esta área, vou criar uma sequência nos dois eixos com um ponto a cada 2 metros, e esta sequência jogarei dentro do expand.grid() que vai criar para nós todas as combinações possíveis entre as duas sequencias. Sendo mais concreto, é como se tivéssemos um ponto a cada dois metros distribuídos de forma regular em uma área de 100 km2.
|
|
>>> x y
>>> 1: 490000 9990000
>>> 2: 490002 9990000
>>> 3: 490004 9990000
>>> 4: 490006 9990000
>>> 5: 490008 9990000
>>> ---
>>> 25009997: 499992 10000000
>>> 25009998: 499994 10000000
>>> 25009999: 499996 10000000
>>> 25010000: 499998 10000000
>>> 25010001: 500000 10000000
Acabei criando muitos pontos (25 010 001), na verdade, não precisaremos de todos eles, pois temos somente 65 845 árvores, ou seja, somente 0.26% dos pontos serão ocupados. Faremos essa alocação de forma aleatória dentro deste espaço virtual que acabamos de criar.
Vamos juntar as duas coisas
|
|
>>> especie altura x y
>>> 1: B 3.7080 496000 9995152
>>> 2: B 2.7710 495730 9995552
>>> 3: D 4.8520 494378 9995186
>>> 4: C 2.7330 499454 9998076
>>> 5: B 2.8380 497760 9999714
>>> ---
>>> 65841: D 3.8050 496288 9991634
>>> 65842: B 2.5280 493400 9991674
>>> 65843: C 0.9099 496014 9997044
>>> 65844: D 3.6320 490392 9998874
>>> 65845: E 1.6730 497718 9995032
Estou juntando duas tabelas, sendo a primeira é aquela com espécie e altura com 65845 linhas e preciso da mesma quantidade de linha daquela tabela com coordenadas, já que eu tenho muitos pontos escolhi a quantidade de linhas que eu preciso de forma aleatória. Teoricamente, eu deveria poder misturar essas árvores fictícias bem nesses 100 km2, veremos isso daqui a pouco.
|
|
>>> especie altura x y
>>> 1: B 3.7080 496000 9995152
>>> 2: B 2.7710 495730 9995552
>>> 3: D 4.8520 494378 9995186
>>> 4: C 2.7330 499454 9998076
>>> 5: B 2.8380 497760 9999714
>>> ---
>>> 65841: D 3.8050 496288 9991634
>>> 65842: B 2.5280 493400 9991674
>>> 65843: C 0.9099 496014 9997044
>>> 65844: D 3.6320 490392 9998874
>>> 65845: E 1.6730 497718 9995032
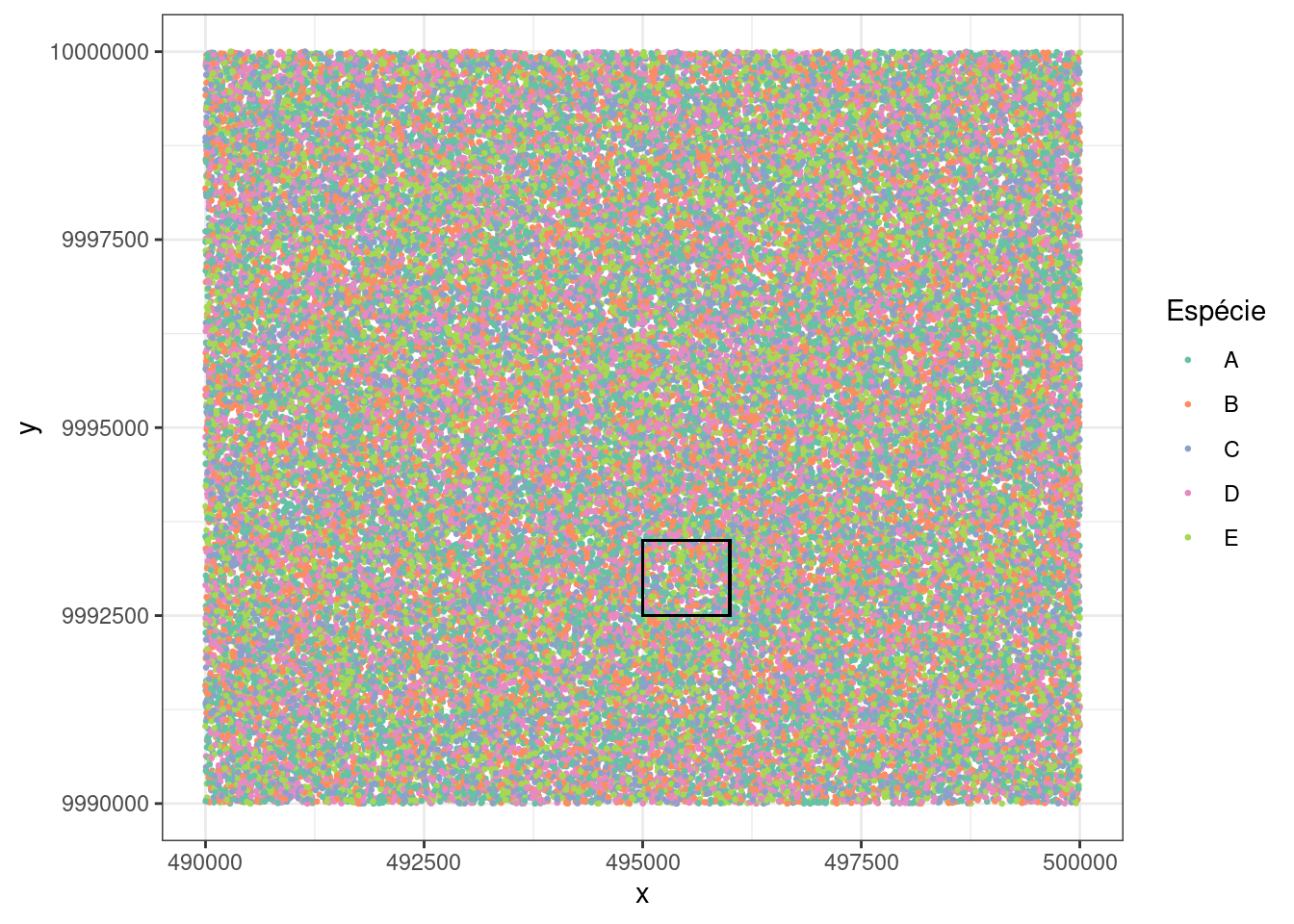
A nossa tabela única está pronta! Vamos explorar duas coisas que assumimos lá em cima, 1) os valores de altura de cada espécie apresentaria uma distribuição normal (faremos uma checagem rápida com histogramas dos dados), 2) conseguiríamos misturar as árvores bem na nossa área virtual (podemos plotar e ver onde caiam).
Plotando os dados!
Histograma dos valores
|
|
Onde caiam os pontos?
|
|
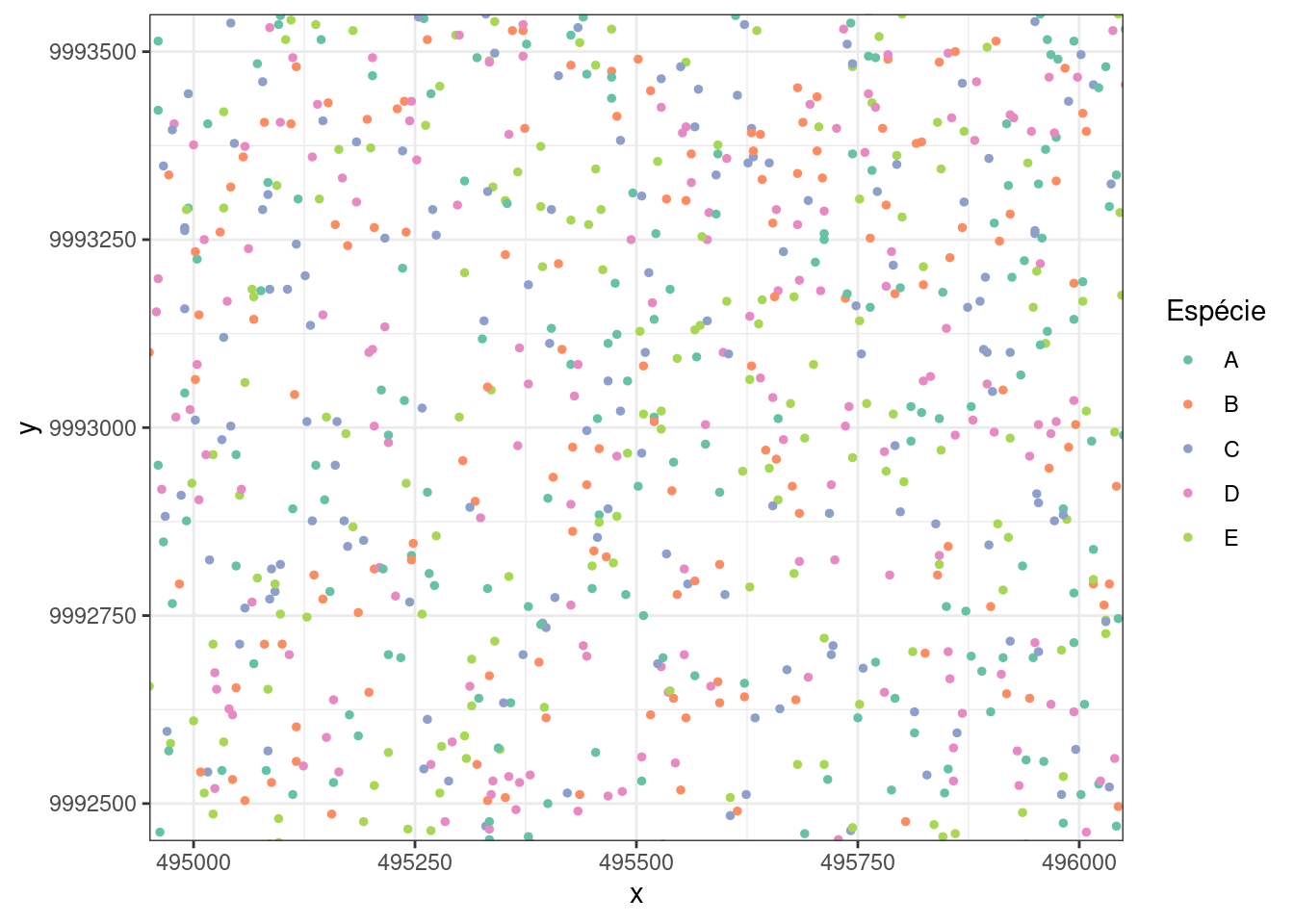
Podemos dar uma aproximação no retangulo em preto
|
|
Salvando em disco
Fico satisfeito com isso, poderíamos parar aqui rs, mas precisamos quebrar essa tabela em várias outras menores. A ideia apresentada foi que os arquivos foram salvos em 10 pastas diferentes. Para isso, precisaremos criar as pastas primeiro e depois encontraremos uma forma de quebrar a tabela em partes sem duplicação e salvá-las nas pastas.
Criando as pastas
Vou criar uma pasta chamada temp_ dentro da minha pasta home (/home/tredgi/temp_), é nela que quero criar essas 10 sub-pastas que chamarei aqui de pasta 001 até 010.
|
|
>>> [1] "/home/tredgi/temp_/pasta_001" "/home/tredgi/temp_/pasta_002"
>>> [3] "/home/tredgi/temp_/pasta_003" "/home/tredgi/temp_/pasta_004"
>>> [5] "/home/tredgi/temp_/pasta_005" "/home/tredgi/temp_/pasta_006"
>>> [7] "/home/tredgi/temp_/pasta_007" "/home/tredgi/temp_/pasta_008"
>>> [9] "/home/tredgi/temp_/pasta_009" "/home/tredgi/temp_/pasta_010"
Criando nomes
Até aqui, temos a tabela única, temos as pastas. Falta quebrar a tabela em várias sub tabelas, cada sub tabela será salvo na pasta escolhida com algum nome. Vamos criar uma função criadora nomes para as sub tabelas.
|
|
>>> [1] "HRGOG7935W" "EHPZQ9011S" "BABFR3718Q" "WEQLB2559A" "HWYZV3960Z"
O padrão escolhido para cada nome é 5LETRAS+4números+1LETRA. Temos uma criadora de nomes que seguira sempre este padrão.
Salvando de fato
Agora, falta encontrar uma forma de pegar partes da tabela, sem repetir a mesma linha. Eu poderia simplesmente ir pegando de tal linha até, tal outra até acabar. Mas eu quero explorar outra forma de fazer isso de forma aleatória (este post vai acabar sendo um post aleatório 😅).
No R conseguimos escolher uma linha em uma tabela com o colchete (ex: minha_tabela[index_linha_desejada, ]). Pode ser uma ou varias linhas em simultâneo. O índex que aqui é o número de linhas pode ser criada com seq_len(nrow(df_x_y)), assim passamos a ter um vetor com todos os números de cada linha da tabela. Trabalhar com este vetor exige menos poder de processamento se comparado com mexer com a tabela toda em um processo iterativo. Criaremos este vetor, penso em deixar entre 65 e 80 tabelas em cada pasta e cada arquivo entre 85 e 95 linhas, também vou querer colocar alguns arquivos com extensão estranha além daqueles .txt. Vamos ver como ficou o código.
|
|
>>> [1] 65845
Note que no começo, o vetor
df_x_y_idtem 65 845 elementos.
|
|
|
|
>>> [1] 744
Depois das iterações, aquele vetor
df_x_y_idcom 65 845 elementos passa a ter somente 744 elementos. Isso porque a cada iteração(linha 19)os valores escolhidos de forma aleatória foram excluídos para não correrem o risco de serem escolhidos de novo.
Então, foi esse o processo de criação de toda a estrutura de pastas e arquivos para a demonstração com o Sell Bash, onde eu realizei o processo inverso do que é feito aqui.
Limpando o disco
Após criar e usar esses arquivos, podemos limpar tudo estando no R mesmo, com essa linha:
|
|
Todo o código!
|
|